2. 贵州省人民医院 耳鼻咽喉科, 贵州 贵阳 550002
2. Department of Otolaryngology, the Guizhou Provincial People's Hospital, Guiyang 550002, China
慢性化脓性中耳炎(chronic suppurative otitis media,CSOM)是一种常见病及多发病,该疾病为中耳黏膜、骨膜或深达骨质的化脓性炎症,重者炎症可深达乳突骨质。本病以患耳长期间歇或持续流脓、鼓膜穿孔及听力下降为特点[1],由于中耳解剖位置特殊性,CSOM可引起颅内外并发症,特别是颈深部感染、脑膜炎、脑脓肿等,严重者可能导致死亡。在组织学上,CSOM中耳黏膜的炎症表现为组织水肿、黏膜下纤维化、血管增生、淋巴细胞、浆细胞和组织细胞浸润。炎症过程通常伴随着黏膜溃疡和血管、成纤维细胞和炎症细胞的增殖,导致肉芽组织的形成。鼓膜穿孔导致患者听力障碍和中耳腔恶臭的分泌物可能会对患者身体和心理造成持久的影响。CSOM也被认为是儿童和年轻患者最常见的耳科疾病[2-3]。
1 CSOM目前在全球范围流行现状根据世界卫生组织(WHO)初步估计,现在全球大约有6 500万至3.3亿人患有CSOM,而每年大约21 000人死于CSOM的并发症。疾病负担最重的是沙哈拉以南的非洲和大洋洲低收入国家,主要是由于营养不良、过度拥挤、卫生条件差、医疗保健不足和反复上呼吸道感染,该疾病在发展中国家的发病率较高,特别是在社会经济水平较低的地区,城市与农村的发病率为1∶2,较贫穷的农村社区发病率最高[4]。对于人群基本属性来讲,女性及年轻患者发病率较高[5-6],但也有研究指出男性患病率高于女性[7],在我国的一项调查中显示55%患者为男性,45%患者为女性[8],这有可能与两性对求医的态度和对疾病的耐受能力有关。由于地区、民族、饮食习惯等差异,CSOM在世界各地的发病率也有所差异,全球总的发病率约为4.76/1 000人,每年新增患者约3 100万人[2],据不同地区的研究报告,在孟加拉国,CSOM的患病率为7.4%~39.5%,在印度为7.8%~16%[9],在中国还没有CSOM确切的患病率[8]。
对于发病年龄来讲,前面讲到CSOM高发年龄段为儿童期,特别是6岁以内比较常见的疾病[10-11],其主要原因为儿童咽鼓管较短、位置较平、软骨较松弛和免疫力低下,各国家地区的差异也比较明显。在中国的一项研究中发现发病率最高的年龄段为51~60岁[8],Mofatteh等[6]发现发病率最高的年龄段为21~30岁,然而,也有部分研究发现发病率最多的年龄段为<20岁的患者[12-13]。
2 CSOM细菌感染的菌种研究 2.1 CSOM常见菌种地域的差异分布CSOM主要是急性中耳炎(acute suppurative otitis media, AOM)迁延不愈的结果[14],而急性感染如何发展为慢性疾病仍然很大程度上不清楚[15],细菌感染是CSOM最常见的一种病因。由于呼吸道黏膜的邻近性,由口咽和鼻咽淋巴组织的上行性感染容易导致中耳反复感染,以及鼓膜穿孔导致持续性耳漏[16]。了解CSOM的流行病学情况、病原微生物和抗生素的敏感性对于根据经验选择最有效的抗生素以及了解感染的一般过程至关重要[17]。另外,我们研究中的优势微生物及其抗生素敏感性也在不断地变化,所以,我们必须对微生物进行持续和定期检测,以指导选择合适的抗生素[18]。
CSOM的微生物群主要包括需氧菌、厌氧菌和真菌等,根据患者的年龄、地理位置、种族和是否有胆脂瘤等并发症,其相应的发病率和菌种有所不同[13, 19]。Xu等[20]认为CSOM和中耳胆脂瘤(middle ear cholesteatoma,MEC)是两种不同的疾病,所以这两种疾病的致病微生物也应该有所差异,通过常规细菌培养发现,CSOM组G+细菌检出率高于MEC组,CSOM组G+细菌检出率低于MEC组。两组主要病原菌为金黄色葡萄球菌、铜绿假单胞菌、变形杆菌和凝固酶阴性葡萄球菌。Xu等[8]对289例CSOM患者中耳腔分泌物进行培养,G+细菌为60.2%,G-细菌为39.8%,以G+细菌占优势,84%的患者为CSOM不伴胆脂瘤,而16%为CSOM伴胆脂瘤。具体细菌来讲,金黄色葡萄球菌为主要的致病菌,约占44.9%,在CSOM伴或不伴胆脂瘤中金黄色葡萄球菌有相似的结果,耐甲氧西林金黄色葡萄球菌(methicillin-resistant staphylococcus aureus,MRSA)占金黄色葡萄球菌总数的18.9%。铜绿假单胞菌是第二常见致病菌,约占16.9%,在CSOM不伴胆脂瘤中约占15.7%,在CSOM伴胆脂瘤中约占23.7%,也有部分为金黄色葡萄球菌、铜绿假单胞菌和阴沟肠杆菌的混合菌。在伴有胆脂瘤的CSOM中,肺炎克雷伯菌是第三常见的细菌,约占7.9%,在无胆脂瘤的CSOM中,凝固酶阴性葡萄球菌是第三常见的致病菌,约占9.6%。金黄色葡萄球菌、铜绿假单胞菌和凝固阴性葡萄球菌是CSOM的3种主要致病菌[8]。另有国内研究,苏州地区中耳炎分泌物细菌培养阳性率为69.4%,其中肺炎链球菌、金黄色葡萄球菌和流感嗜血杆菌为主要的致病菌[15],重庆地区中耳炎分泌物主要菌种为金黄色葡萄球菌、铜绿假单胞杆菌等[21]。从以上几个国内研究表明,金黄色葡萄球菌、铜绿假单胞菌是我国CSOM比较常见的致病菌。
国外一些研究中获得菌种类别与国内有一定的差异。Hiremath等[22]发现,在120例CSOM最常见的需氧菌为铜绿假单胞菌(38.79%),其次为金黄色葡萄球菌(32.75%)。Mittal等[10]发现CSOM主要需氧菌由铜绿假单胞菌、大肠杆菌、金黄色葡萄球菌、化脓性链球菌、奇异变形杆菌、肺炎克雷伯菌组成,然而,厌氧菌由类杆菌、消化链球菌、丙酸杆菌组成。
2.2 CSOM中非常见微生物菌种 2.2.1 多重耐药菌多重耐药菌是在抗生素不合理使用情况下出现的产物,一些多重耐药菌在患者耳部脓液中被发现,这将是对全球的医疗管理的一个重大挑战,另一方面,多重耐药菌的出现将增加CSOM的复发率[23-24]。MRSA是多重耐药菌中最常见的一种,在韩国的一项研究中MRSA的分离率为22.8%。美国的一项调查提示,由社区获得性MRSA感染继发的CSOM从1998年的0.7%增加到2006年的11.4%[25]。印度的一项研究提示,在CSOM中MRSA感染占金黄色葡萄球菌总数的34.3%[26]。
2.2.2 真菌感染真菌性感染在一些病例中也时常被发现。关于CSOM继发性真菌感染的确切患病率的信息明显缺乏。中耳真菌感染通常被误诊为细菌感染,因此患者在最初阶段会被告知使用抗生素滴耳剂。只有当抗生素滴耳剂不能改善症状时才考虑到有真菌感染的可能[27],使用抗生素出现菌群失调后导致抑制细菌菌群和随后出现的真菌菌群,可能会增加真菌重复感染的发生率,甚至毒性较低的真菌也可能变成致病菌。此外,真菌从CSOM患者中分离的比率也存在很大程度的差异[28]。在中国,上海地区真菌性中耳炎中,女性发病率高于男性,大约女∶男为2∶1,主要菌种为曲霉菌和白色念珠菌[27, 29-30]。印度医生Philip等[29]对怀疑真菌性中耳炎15例患者接受手术后病理明确真菌的存在,主要菌种为毛霉菌、曲霉菌及白色念珠菌[31]。真菌感染很难根治,其最重要的原因是可能形成的生物膜导致的复发,同时已经证实念珠菌可以形成生物膜[30, 32]。
2.2.3 海藻希氏菌海藻希氏菌是一种广泛分布于环境中的兼性厌氧菌及腐生的革兰氏阴性杆菌,是海洋微生物群中的一员,是一种罕见的人类病原体,在温带气候地区流行率很高,如美国、南非、澳大利亚、亚洲和南欧[33-34]。直接接触海水或食用受污染的海鲜是众所周知的感染风险因素[35-38]。据报道,海藻希氏菌引起了广泛的临床疾病,如皮肤和软组织感染、菌血症、胃肠炎、小脑脓肿、耳和眼部感染、感染性心内膜炎和心包炎。土耳其的一些报告与该细菌相关的病例有肺炎、小脑脓肿、脑膜炎和伤口感染[39-40]。在丹麦的一项研究中,最常见的由海藻希氏菌引起中耳感染是55例患者中有47例在接触海水后出现CSOM的症状[41],这也是海藻希氏菌作为CSOM非常见细菌的有力证据。
2.2.4 肠球菌由于地域及生活习惯的差异,一些远离耳部的器官常见细菌也在患者耳部脓液中被发现。对于肠球菌的检出,首先考虑到是否为标本污染,但是后来发现也有可能与该地区一些习俗有关,部分患者为了减少耳部脓性分泌物,患者偶尔会使用鸽子的粪便填满于自己的外耳道。同时该地区的一些患者还使用蟑螂膏、棕榈油、甜橄榄油、缝纫机油或母乳来试图减少耳部脓性分泌物,上述的各种习俗可能解释中耳脓性分泌物中肠球菌的发现。因为局部地区肠道菌群在CSOM患者中发现也间接说明该地区卫生模式较差,使得人类有更多的机会接触动物和人类的粪便,因此我们在重视临床管理的同时还要加强生活环境的管理[42]。
既往观点认为正常人体的中耳腔都是无菌的[43-44]。目前对健康和疾病状态中耳和乳突黏膜微生物学的了解主要来自细菌培养研究。由于某些细菌无法在标准培养基中生长,这些结果不可避免地会产生偏差[45]。CSOM的致病菌可能来自中耳或乳突腔内的共生细菌,在健康对照组的中耳腔内也检测出了潜在的病原体,比如:葡萄球菌、假单胞菌、链球菌和莫拉菌,这支持了中耳腔或乳突腔内常驻微生物群是潜在的内部感染源的观点。在人体的其他黏膜部位,共生生物在防御机会性致病菌入侵方面发挥重要作用。例如在肠道中,细菌之间的交流和调节可以对细菌的生长产生积极或消极的影响。这种相互作用似乎是通过细菌释放信号分子并被宿主细胞重新吸收而介导的。类似的相互作用可能发生在中耳,但仍有待证明[46-47]。由于某些细菌无法在标准培养基中生长,相比之下分子生物学技术能够更准确地评估生长在黏膜上的微生物群落。现在大部分医院常规实验室诊断通常会选择性地培养已知病原体,所以其他病原体可能会被忽视。正因为考虑到许多人类微生物是不可培养的或极其难以生长的,所以选择分子工具能够更好地了解样本中细菌群落的组成。
3 细菌生物膜在CSOM中作用 3.1 细菌生物膜概述细菌生物膜是一种细菌群落,其形成是原核生物在恶劣环境中生存的关键组成部分。这些群落是细菌植入一种被称为细胞外聚合物(extracellular polymeric substances, EPS)的营养基质中形成的[48]。细菌生物膜是由细胞外DNA、蛋白质和多糖等胞外聚合物质组成的微生物群落。生物膜形成是一种毒性机制,在健康宿主和有潜在疾病的宿主中建立持续感染[49-51]。早在1683年,Antoni利用显微镜在自己的牙齿上发现了生物膜,但是并未引起重视。直到20世纪70年代初期观察到囊性纤维化患者持续感染与细菌聚集之间的联系,医学微生物学家才对生物膜的生活方式产生兴趣,并对其进一步研究[52]。
生物膜的形成需要几个重要的步骤。首先,在组织表面一些少量的有机物形成一个中和层,这些中和层更容易使细菌附着。然后,细菌接触到中和层,使细菌与中和层不可逆的结合。最后,由于细菌不断聚集,EPS开始形成,随着细菌细胞的生长和EPS形成增加,生物膜开始成熟。成熟的生物膜EPS含量远高于细菌含量,且大部分细菌处于休眠期。一旦生物膜成熟,便可释放细菌导致局部或全身炎症形成[53-54]。
3.2 细菌生物膜的致病机制由于多种因素,生物膜中的细菌对环境压力、抗菌治疗和宿主免疫具有更强的抵抗力。EPS或生物膜基质为生物膜提供了结构完整性,并作为物理屏障将生物膜内的细胞与细胞外环境分离[55-56]。生物膜的结构在生物膜内部的营养与垃圾交换起到重要的作用[57]。生物膜内的细菌生长速率的变化和代谢的停止增加了对抗生素的耐药性[58]。除了基质提供的保护外,生物膜中的细菌还可以采用其他生存策略来逃避宿主的防御系统。细菌通过处于休眠状态和躲避免疫系统,它们可能会造成局部组织损伤,随后引起急性感染。在生物膜内,细菌通过代谢改变、基因表达和蛋白质生产来适应缺氧和营养缺乏的环境,这可以导致代谢率和细胞分裂率降低[59]。此外,还能通过灭活抗菌靶点或减少抗菌素干扰细胞功能,使生物内的细菌对抗菌治疗更具耐药性。在生物膜感染期间,机体还可能同时激活先天和后天性宿主免疫反应,但这两种免疫反应都不能消除生物膜病原体,反而加速了相关组织的损伤[60]。
人体约80%的慢性感染是由生物膜导致的[61],且生物膜在耳鼻咽喉科感染中的重要性日益明显[62]。生物膜的形成是细菌在中耳内持久性的一个主要因素,抗菌素耐药性的增加归因于细菌代谢的变化和生长速度的下降[58]。营养供应和应激的差异可能会导致铁获取、氧化应激和生物膜内代谢蛋白的变化。此外,黏膜表面蛋白表达和生物膜形成的差异降低了宿主对细菌的反应和识别,并可能改变宿主固有免疫防御的有效性[63-64]。逆转录聚合酶链反应显示,在中耳腔积液阴性培养患者中存在代谢活菌,这验证了生物膜是慢性中耳炎发病机制的一部分假设[65]。后来的研究发现,通过扫描电子显微镜和共聚焦激光扫描显微镜等多种方式,在人类和动物中耳炎模型均发现生物膜,所以生物膜通常被认为是中耳炎发病机制的一部分[66-67]。
3.3 细菌生物膜在中耳慢性炎症疾病中的研究人体的中耳腔并不是一个密闭的空间,正常的中耳腔可以通过咽鼓管与鼻腔、咽腔相通,因此人体中耳腔中会有大量常驻菌存在,但在某些病理情况下(外伤、鼓膜穿孔等)导致中耳腔内急慢性炎症形成,在一些体质差的患者或治疗不彻底的情况下可能会导致生物膜的形成,这在一些CSOM患者中耳脓性分泌物[9]或病理组织[68-71]中得以证实。侯炜等[72]为了研究细菌生物膜,利用铜绿假单胞菌构建小鼠CSOM模型,并发现在构建模型6 d后所有中耳标本黏膜表面均出现具有典型特征的细菌生物膜,表面被胞外多糖包裹,扫描电镜还观察到细菌生物膜呈“蘑菇状”的三维结构及其之间的水通道,同时还发现随着生物膜的生长和成熟,中耳炎的症状也逐渐加重。因此,在所有的CSOM和耳部持续流脓超过14 d的患者中耳腔均有可能形成生物膜[55]。细菌生物膜可作为一个细菌库,然后细菌从生物膜中扩散,这也可能是CSOM复发和形成慢性感染的原因。
对于生物膜导致CSOM复发和形成慢性感染的机制仍不完全清楚,目前主要认为CSOM可能的机制为生物膜的形成与持续感染和抗生素治疗效果降低有关,且生物膜已被证明有助于细菌、酵母和丝状真菌的耐药性,并且可能是造成最低抑菌浓度数据和临床结果之间差异的一个因素[73],同时生物膜内细菌被包裹在聚合物基质中,降低了它们对抗菌药物和免疫防御的敏感性,这种固有的抗菌药物耐药性为抗生素治疗和宿主防御提供了额外的耐药性。此外,在感染过程中,起源于生物膜的细菌以浮游细胞的形成扩散,导致感染的进展[59, 74]。生物膜内的持久性细胞在降低抗生素作用方面起到重要的作用,持久性细胞是CSOM顽固性感染的主要驱动力,该细胞代谢不活跃,能够逃避抗生素的攻击[75],同时持久性细胞对抗生素的应答能力不是可遗传的特征,而是随机的表型分化[76]。持久性细胞的产生主要是由于生物膜的低营养和低氧水平,正是由于持久性细胞代谢活性低,故该细胞在抗生素的作用损伤后仍然能够得到很好的恢复[76],所以认为CSOM产生的主要问题是生物膜内的持久性细胞,而不是生物膜本身,因此,CSOM应被认为是一种由持久性细胞引起的疾病[77]。另外关于细菌生物膜加重CSOM感染的机制可能因为细菌生物膜胞外多糖基质的主要成分藻酸盐,这种藻酸盐具有明显的抗原性,再和细菌的各种抗原叠加,多种抗原共同刺激机体产生大量特异性抗体。由于胞外多糖基质的屏障作用,使得抗体不能顺利进入细菌生物膜内而不能消灭其内部的细菌,而在表面形成免疫复合物,趋化中性粒细胞与免疫复合物的FC端结合,激活、释放出各种酶类物质,溶解破坏细菌生物膜周围的正常组织,导致发生免疫病理变化,从而加重CSOM的感染[78]。
细菌生物膜在CSOM的致病机制已被重视,生物膜对抗生素和其他抗菌化合物具有耐药性[79-81],因此很难根除,并且导致中耳腔反复感染。另外,生物膜附着在中耳受损的组织上,例如:中耳腔暴露性骨炎、中耳腔黏膜溃疡,或者是耳科手术植入物(鼓膜置管等),进一步加重了中耳炎的反复[82]。虽然生物膜在CSOM中已被证实,但它们在疾病中的具体病理生理学作用尚不明确[83],需要我们进一步去探索。
4 CSOM微生物学常见分析方法 4.1 基于培养技术基于培养技术的分析研究是根据不同微生物的生长特性选择不同的培养条件进行而达到分离的目的,然后根据微生物的形态学和生化方法来鉴定物种及药物敏感试验,这种技术也是目前绝大部分医院进行感染病原学的诊断方法。然而体外培养很难模拟微生物的自然生存条件,就微生物比较丰富的肠道而言,目前为止仅仅培养了30%的肠道微生物,但并不是指大约70%的肠道菌群无法培养,只是表明非常难以确定这些微生物最优的生长条件[84]。另外,培养技术还可能存在以下的缺点:①某些细菌的培养周期长,甚至达1周以上;②厌氧菌一般难以培养;③菌落形成不良可能与患者之前使用抗生素有关[85]。
4.2 16S rRNA扩增子测序16S rRNA扩增子测序是以细菌为研究对象,通过直接扩增环境微生物总DNA的特定区域,研究微生物分布、丰度变化及样本间微生物群落组成差异情况(简要流程见图 1)。16S rRNA大约1540bp,基因序列长短适中,含有10个保守区和9个可变区(V1~V9),变异频率低,占细菌总RNA的80%以上,所以16S rRNA扩增子测序被用作最常用的微生物鉴定技术[86]。目前,大规模的细菌16S rRNA基因分析都是基于对细菌16S rRNA基因可变区测序,包括V2、V3、V4、V1-V2、V1-V3、V2-V3、V2-V4、V3-V4、V4-V5、V3-V6、V7-V9均采用第二代测序技术进行测序,此前由罗氏公司提供,后来由Illumina公司提供。然而,由于测序区域的不同,测序结果也有可能出现偏差[87]。Illumina MiSeq测序是目前应用最广泛的细菌16S rRNA基因扩增子测序平台。它最新的化学结构允许测序300 bp双向PCR片段完全重叠,适合于单可变区和细菌16S rRNA V1-V2基因区域的测序,需要全序列重叠[87]。然而,第二代测序的短读方法因使用的可变区域而存在偏差,不能提供低于细菌属水平的有效分辨率,限制了微生物生态学研究。Gosiewski等[88]和Gyarmati等[89]利用16S rRNA基因扩增分析血液样本查找可疑的致病菌,但这两项研究都无法在物种层面上找到令人信服的致病细菌,可能是16S rRNA仅分析了V1-V3或V3-V4区扩增子导致reads信息不足而引起。
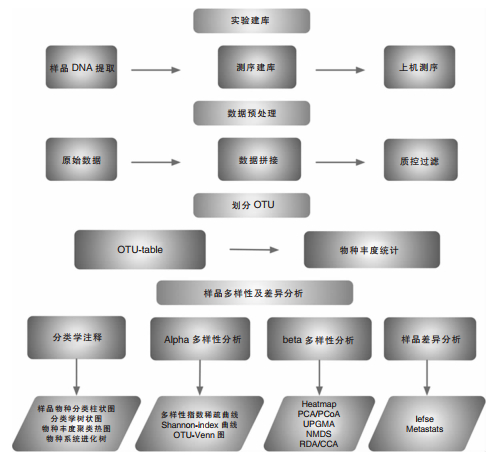
|
| 注:OTU(操作分类单元);PCA分析(主成分分析);PCoA分析(主坐标分析);NMDS分析(非度量多维标定法);UPGMA分析(非加权组平均法);lefse分析(线性判别分析效应大小);RDA/CCA分析(相关性与关联分析)。下同。 图 1 16S rRNA扩增子测序简要流程 |
16S rRNA扩增子测序长期以来一直是微生物组鉴定的首选方法。然而,16S rRNA测序通常只有部分可变区域被测序很少能提供物种水平的分辨率,不能直接评估其他类群(如病毒和真菌)或功能基因含量。此外,部分16S rRNA序列读取可能无法提供分辨16S rRNA高度同源物种所需的分辨率[90]。尽管利用基于16S rRNA的数据已经取得了突破性的发现,但物种水平的具体分辨率对于了解微生物已经变得越来越重要[91-92]。该方法具有引物偏差和缺乏特异性的缺点,但其在大样品量、低成本和高灵敏度方面却占有较大的优势。
4.3 宏基因组测序技术宏基因组测序(metagenomic sequencing,MGS),利用二代/三代高通量测序技术对环境样本中全部微生物基因组DNA进行测定,以分析微生物群体的遗传多样性和丰度,解读微生物群体的基因组成及功能多样性,探索微生物与环境/宿主之间的相互作用关系,发掘具有新的特定功能的基因(简要实验流程见图 2,简要分析流程见图 3)。新一代宏基因组测序通过高通量测序和自动化生物信息分析,对标本中所有基因组对应的已知微生物均能在短时间内(1~6 d)进行鉴定。MGS已被用于检测脑脊液[93-94]、血液[95-96]、尿液[97]、支气管肺泡灌洗液样本[98]和骨关节感染[99]等样本中的病原体。MGS相对于常规培养有以下优势:①对病原体的总体检出率明显高于常规培养;②MGS受抗菌药物治疗影响较小,表明MGS具有足够的灵敏度,可以检测标本中低水平的微生物或残留核酸;③MGS在接受标本后几天内就可以获得最终报告[99]。但是由于MGS价格较昂贵,所以在医院大规模开展存在一定的难度。
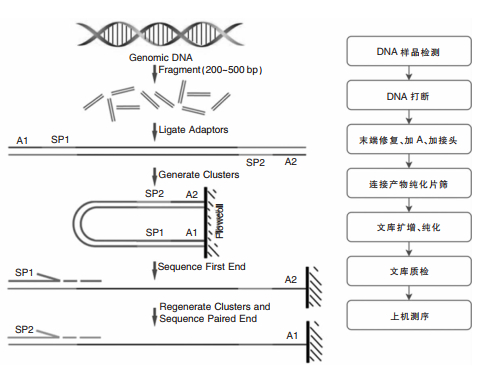
|
| 图 2 宏基因组测序技术简要实验流程 |
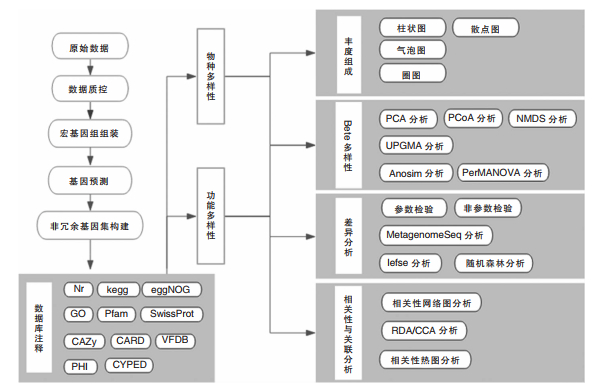
|
| 注:Anosim分析(相似性分析);PerMANOVA分析(置换多元方差分析)。 图 3 宏基因组测序技术简要分流流程 |
细菌感染在CSOM的致病机制中已被充分重视,在治疗中使用的广谱抗生素对共生细菌群落产生不利影响,通过消除相互竞争的共生细菌,可能为病原体提供竞争优势,所以我们应该在明确致病菌情况下合理、合规使用抗生素,以达到治疗效果。但CSOM的中耳腔微环境是多种细菌,甚至真菌的集合,使用目前的细菌培养技术可能不能获得满意的细菌谱,也会丢失一些重要的细菌,故需要利用新一代的分子生物学技术来获取中耳腔的微生态情况,这样不仅能获得满意的细菌谱,提供准确的治疗方案,减少因大量广谱抗生素使用带来的细菌耐药问题,也能够丰富当地CSOM感染细菌谱。但是利用分子生物学技术来检测细菌,价格昂贵,在我国部分地区常规开展存在困难,故在探索CSOM中耳腔的微生态道路上仍然任重道远。
| [1] |
Molla R, Tiruneh M, Abebe W, et al. Bacterial profile and antimicrobial susceptibility patterns in chronic suppurative otitis media at the University of Gondar Comprehensive Specialized Hospital, Northwest Ethiopia[J]. BMC Res Notes, 2019, 12(1): 414.
[DOI]
|
| [2] |
Monasta L, Ronfani L, Marchetti F, et al. Burden of disease caused by otitis media: systematic review and global estimates[J]. PLoS One, 2012, 7(4): e36226.
[DOI]
|
| [3] |
Li MG, Hotez PJ, Vrabec JT, et al. Is chronic suppurative otitis media a neglected tropical disease?[J]. PLoS Negl Trop Dis, 2015, 9(3): e0003485..
[DOI]
|
| [4] |
Wang J, Chen B, Xu M, et al. Etiological factors associated with chronic suppurative otitis media in a population of Han adults in China[J]. Acta Otolaryngol, 2016, 136(10): 1024-1028.
[DOI]
|
| [5] |
Shaheen MM, Nahar S. Comparison of chronic suppurative otitis media in rural and urban primary school children in Bangladesh[J]. J Laryngol Otol, 2014, 128(6): 499-503.
[DOI]
|
| [6] |
Mofatteh MR, Shahabian Moghaddam F, Yousefi M, et al. A study of bacterial pathogens and antibiotic susceptibility patterns in chronic suppurative otitis media[J]. J Laryngol Otol, 2018, 132(1): 41-45.
[DOI]
|
| [7] |
Udden F, Filipe M, Reimer A, et al. Aerobic bacteria associated with chronic suppurative otitis media in Angola[J]. Infect Dis Poverty, 2018, 7(1): 42.
[DOI]
|
| [8] |
Xu J, Du Q, Shu Y, et al. Bacteriological profile of chronic suppurative otitis media and antibiotic susceptibility in a tertiary care hospital in Shanghai, China[J]. Ear Nose Throat J, 2021, 100(9): NP391-NP396.
[DOI]
|
| [9] |
Jensen RG, Johansen HK, Bjarnsholt T, et al. Recurrent otorrhea in chronic suppurative otitis media: is biofilm the missing link?[J]. Eur Arch Otorhinolaryngol, 2017, 274(7): 2741-2747.
[DOI]
|
| [10] |
Mittal R, Lisi CV, Gerring R, et al. Current concepts in the pathogenesis and treatment of chronic suppurative otitis media[J]. J Med Microbiol, 2015, 64(10): 1103-1116.
[DOI]
|
| [11] |
Ayaz Z, Taj B, Yaseen MS, et al. Causality of chronic suppurative otitis media: an observational study[J]. Cureus, 2020, 12(8): e9832.
|
| [12] |
Abraham ZS, Ntunaguzi D, Kahinga AA, et al. Prevalence and etiological agents for chronic suppurative otitis media in a tertiary hospital in Tanzania[J]. BMC Res Notes, 2019, 12(1): 429.
[DOI]
|
| [13] |
Chirwa M, Mulwafu W, Aswani JM, et al. Microbiology of chronic suppurative otitis media at Queen Elizabeth Central Hospital, Blantyre, Malawi: A cross-sectional descriptive study[J]. Malawi Med J, 2015, 27(4): 120-124.
|
| [14] |
Adoga A, Nimkur T, Silas O. Chronic suppurative otitis media: Socio-economic implications in a tertiary hospital in Northern Nigeria[J]. Pan Afr Med J, 2010, 4: 3.
|
| [15] |
Ding Y, Geng Q, Tao Y, et al. Etiology and epidemiology of children with acute otitis media and spontaneous otorrhea in Suzhou, China[J]. Pediatr Infect Dis J, 2015, 34(5): e102-106.
|
| [16] |
Khattak SF, Sheikh NA, Aleem A, et al. Microbiological profile from middle ear and nasopharynx in patients suffering from chronic active mucosal otitis media[J]. J Ayub Med Coll Abbottabad, 2017, 29(4): 610-613.
|
| [17] |
Brook I. The role of anaerobic bacteria in chronic suppurative otitis media in children: implications for medical therapy[J]. Anaerobe, 2008, 14(6): 297-300.
[DOI]
|
| [18] |
Yeo SG, Park DC, Hong SM, et al. Bacteriology of chronic suppurative otitis media--a multicenter study[J]. Acta Otolaryngol, 2007, 127(10): 1062-1067.
[DOI]
|
| [19] |
Khatun MR, Alam KMF, Naznin M, et al. Microbiology of chronic suppurative otitis media: an update from a tertiary care hospital in Bangladesh[J]. Pak J Med Sci, 2021, 37(3): 821-826.
|
| [20] |
Xu F, Kong W, Peng J, et al. Analysis of main pathogenic bacteria and drug sensitivity in patients with chronic suppurative otitis media and middle ear cholesteatoma in China[J]. Biotechnol Lett, 2020, 42(8): 1559-1566.
[DOI]
|
| [21] |
孟德静, 吴兴礼, 李艮平, 等. 重庆地区慢性化脓性中耳炎分泌物培养及药敏试验结果分析[J]. 中国耳鼻咽喉颅底外科杂志, 2021, 27(06): 661-665. [URI]
|
| [22] |
Hiremath B, Mudhol RS, Vagrali MA. Bacteriological profile and antimicrobial susceptibility pattern in chronic suppurative otitis media: A 1-year cross-sectional study[J]. Indian J Otolaryngol Head Neck Surg, 2019, 71(Suppl 2): 1221-1226.
|
| [23] |
Choi HG, Park KH, Park SN, et al. The appropriate medical management of methicillin-resistant Staphylococcus aureus in chronic suppurative otitis media[J]. Acta Otolaryngol, 2010, 130(1): 42-46.
[DOI]
|
| [24] |
Park MK, Jung MH, Kang HJ, et al. The changes of MRSA infections in chronic suppurative otitis media[J]. Otolaryngol Head Neck Surg, 2008, 139(3): 395-398.
[DOI]
|
| [25] |
Emmett SD, Kokesh J, Kaylie D. Chronic ear disease[J]. Med Clin North Am, 2018, 102(6): 1063-1079.
[DOI]
|
| [26] |
Rath S, Das SR, Padhy RN. Surveillance of bacteria Pseudomonas aeruginosa and MRSA associated with chronic suppurative otitis media[J]. Braz J Otorhinolaryngol, 2017, 83(2): 201-206.
[DOI]
|
| [27] |
Jia X, Liang Q, Chi F, et al. Otomycosis in Shanghai: aetiology, clinical features and therapy[J]. Mycoses, 2012, 55(5): 404-409.
[DOI]
|
| [28] |
Prakash R, Juyal D, Negi V, et al. Microbiology of chronic suppurative otitis media in a tertiary care setup of uttarakhand state, India[J]. N Am J Med Sci, 2013, 5(4): 282-287.
[DOI]
|
| [29] |
Philip A, Thomas R, Job A, et al. Effectiveness of 7.5 percent povidone iodine in comparison to 1 percent clotrimazole with lignocaine in the treatment of otomycosis[J]. ISRN Otolaryngol, 2013, 239730.
|
| [30] |
Robbins N, Uppuluri P, Nett J, et al. Hsp90 governs dispersion and drug resistance of fungal biofilms[J]. PLoS Pathog, 2011, 7(9): e1002257.
[DOI]
|
| [31] |
Punia RS, Singhal SK, Kundu R, et al. Fungal suppurative otitis media (Histopathology) among patients in North India[J]. Head Neck Pathol, 2019, 13(2): 149-153.
[DOI]
|
| [32] |
Kakeya H, Imamura Y, Miyazaki T, et al. [Chronic fungal infection, up-to-date][J]. Kansenshogaku Zasshi, 2011, 85(4): 333-339.
[DOI]
|
| [33] |
Janda JM, Abbott SL. The genus Shewanella: from the briny depths below to human pathogen[J]. Crit Rev Microbiol, 2014, 40(4): 293-312.
[DOI]
|
| [34] |
Jacob-Kokura S, Chan CY, Kaplan L. Bacteremia and empyema caused by Shewanella algae in a trauma patient[J]. Ann Pharmacother, 2014, 48(1): 128-136.
[DOI]
|
| [35] |
Takata T, Chikumi H, Morishita S, et al. Shewanella algae bacteremia in an end-stage renal disease patient: a case report and review of the literature[J]. Intern Med, 2017, 56(6): 729-732.
[DOI]
|
| [36] |
Srinivas J, Pillai M, Vinod V, et al. Skin and soft tissue infections due to Shewanella algae - an emerging pathogen[J]. J Clin Diagn Res, 2015, 9(2): DC16-20.
|
| [37] |
Bravenec CA, Pandit RT, Beaver HA. Shewanella algae keratitis[J]. Indian J Ophthalmol, 2019, 67(1): 148-150.
[DOI]
|
| [38] |
Bernshteyn M, Ashok Kumar P, Joshi S. Shewanella algae - a novel organism causing bacteremia: a rare case and literature review[J]. Cureus, 2020, 12(9): e10676.
|
| [39] |
Patel R, Abraham A, Thomas J, et al. A rare case of pneumonia caused by Shewanella putrefaciens[J]. Case Rep Med, 2012, 2012: 597301.
|
| [40] |
Bulut C, Ertem GT, Gokcek C, et al. A rare cause of wound infection: Shewanella putrefaciens[J]. Scand J Infect Dis, 2004, 36(9): 692-694.
[DOI]
|
| [41] |
Holt HM, Gahrn-Hansen B, Bruun B. Shewanella algae and Shewanella putrefaciens: clinical and microbiological characteristics[J]. Clin Microbiol Infect, 2005, 11(5): 347-352.
[DOI]
|
| [42] |
Katsarolis I, Poulakou G, Analitis A, et al. Risk factors for nasopharyngeal carriage of drug-resistant Streptococcus pneumoniae: data from a nation-wide surveillance study in Greece[J]. BMC Infect Dis, 2009, 9: 120.
[DOI]
|
| [43] |
Westerberg BD, Kozak FK, Thomas EE, et al. Is the healthy middle ear a normally sterile site?[J]. Otol Neurotol, 2009, 30(2): 174-177.
[DOI]
|
| [44] |
Chang J, Lee SH, Choi J, et al. Nasopharynx as a microbiologic reservoir in chronic suppurative otitis media: preliminary study[J]. Clin Exp Otorhinolaryngol, 2011, 4(3): 122-125.
[DOI]
|
| [45] |
Stewart EJ. Growing unculturable bacteria[J]. J Bacteriol, 2012, 194(16): 4151-4160.
[DOI]
|
| [46] |
Vogt SL, Pena-Diaz J, Finlay BB. Chemical communication in the gut: effects of microbiota-generated metabolites on gastrointestinal bacterial pathogens[J]. Anaerobe, 2015, 34: 106-115.
[DOI]
|
| [47] |
Thompson JA, Oliveira RA, Xavier KB. Chemical conversations in the gut microbiota[J]. Gut Microbes, 2016, 7(2): 163-170.
[DOI]
|
| [48] |
Hall-Stoodley L, Costerton JW, Stoodley P. Bacterial biofilms: from the natural environment to infectious diseases[J]. Nat Rev Microbiol, 2004, 2(2): 95-108.
[DOI]
|
| [49] |
Gunn JS, Bakaletz LO, Wozniak DJ. What's on the Outside Matters: The Role of the extracellular polymeric substance of Gram-negative biofilms in evading host immunity and as a target for therapeutic intervention[J]. J Biol Chem, 2016, 291(24): 12538-12546.
[DOI]
|
| [50] |
Schilcher K, Horswill AR. Staphylococcal biofilm development: structure, regulation, and treatment strategies[J]. Microbiol Mol Biol Rev, 2020, 84(3): e00026-19.
[DOI]
|
| [51] |
Roy R, Tiwari M, Donelli G, et al. Strategies for combating bacterial biofilms: a focus on anti-biofilm agents and their mechanisms of action[J]. Virulence, 2018, 9(1): 522-554.
[DOI]
|
| [52] |
Hoiby N. A short history of microbial biofilms and biofilm infections[J]. APMIS, 2017, 125(4): 272-275.
[DOI]
|
| [53] |
Post JC, Hiller NL, Nistico L, et al. The role of biofilms in otolaryngologic infections: update 2007[J]. Curr Opin Otolaryngol Head Neck Surg, 2007, 15(5): 347-351.
[DOI]
|
| [54] |
Vlastarakos PV, Nikolopoulos TP, Maragoudakis P, et al. Biofilms in ear, nose, and throat infections: how important are they?[J]. Laryngoscope, 2007, 117(4): 668-673.
[DOI]
|
| [55] |
Ehrlich GD, Veeh R, Wang X, et al. Mucosal biofilm formation on middle-ear mucosa in the chinchilla model of otitis media[J]. JAMA, 2002, 287(13): 1710-1715.
[DOI]
|
| [56] |
Hall-Stoodley L, Hu FZ, Gieseke A, et al. Direct detection of bacterial biofilms on the middle-ear mucosa of children with chronic otitis media[J]. JAMA, 2006, 296(2): 202-211.
[DOI]
|
| [57] |
Stewart PS. Mini-review: convection around biofilms[J]. Biofouling, 2012, 28(2): 187-198.
[DOI]
|
| [58] |
Nguyen D, Joshi-Datar A, Lepine F, et al. Active starvation responses mediate antibiotic tolerance in biofilms and nutrient-limited bacteria[J]. Science, 2011, 334(6058): 982-986.
[DOI]
|
| [59] |
Hall-Stoodley L, Stoodley P. Evolving concepts in biofilm infections[J]. Cell Microbiol, 2009, 11(7): 1034-1043.
[DOI]
|
| [60] |
Moser C, Pedersen HT, Lerche CJ, et al. Biofilms and host response - helpful or harmful[J]. APMIS, 2017, 125(4): 320-338.
[DOI]
|
| [61] |
Mah TF. Biofilm-specific antibiotic resistance[J]. Future Microbiol, 2012, 7(9): 1061-1072.
[DOI]
|
| [62] |
Saunders J, Murray M, Alleman A. Biofilms in chronic suppurative otitis media and cholesteatoma: scanning electron microscopy findings[J]. Am J Otolaryngol, 2011, 32(1): 32-37.
[DOI]
|
| [63] |
Ahearn CP, Gallo MC, Murphy TF. Insights on persistent airway infection by non-typeable Haemophilus influenzae in chronic obstructive pulmonary disease[J]. Pathog Dis, 2017, 75(4): ftx042.
|
| [64] |
Brockman KL, Azzari PN, Branstool MT, et al. Epigenetic regulation alters biofilm architecture and composition in multiple clinical isolates of Nontypeable Haemophilus influenzae[J]. mBio, 2018, 9(5): e01682-18.
[DOI]
|
| [65] |
Fergie N, Bayston R, Pearson JP, et al. Is otitis media with effusion a biofilm infection?[J]. Clinical Otolaryngology and Allied Sciences, 2004, 29(1): 38-46.
[DOI]
|
| [66] |
Hall-Stoodley L, Hu FZ, Gieseke A, et al. Direct detection of bacterial biofilms on the middle-ear mucosa of children with chronic otitis media[J]. JAMA, 2006, 296(2): 202-211.
[DOI]
|
| [67] |
Ehrlich GD. Mucosal biofilm formation on middle-ear mucosa in the chinchilla model of otitis media[J]. JAMA, 2002, 287(13): 1710-1715.
[DOI]
|
| [68] |
Lee MR, Pawlowski KS, Luong A, et al. Biofilm presence in humans with chronic suppurative otitis media[J]. Otolaryngol Head Neck Surg, 2009, 141(5): 567-571.
[DOI]
|
| [69] |
Hoa M, Syamal M, Schaeffer MA, et al. Biofilms and chronic otitis media: an initial exploration into the role of biofilms in the pathogenesis of chronic otitis media[J]. Am J Otolaryngol, 2010, 31(4): 241-245.
[DOI]
|
| [70] |
Kim SH, Kim MG, Kim SS, et al. Change in detection rate of Methicillin-Resistant Staphylococcus aureus and Pseudomonas aeruginosa and their antibiotic sensitivities in patients with chronic suppurative otitis media[J]. J Int Adv Otol, 2015, 11(2): 151-156.
[DOI]
|
| [71] |
Yadav MK, Chae SW, Go YY, et al. In vitro Multi-species biofilms of Methicillin-Resistant Staphylococcus aureus and Pseudomonas aeruginosa and their host interaction during in vivo colonization of an otitis media rat model[J]. Front Cell Infect Microbiol, 2017, 7: 125.
|
| [72] |
侯炜, 李晓, 肖红俊. 慢性化脓性中耳炎大鼠中耳黏膜的细菌生物膜形成特点及意义[J]. 临床耳鼻咽喉头颈外科杂志, 2012, 26(1): 30-33. [CNKI]
|
| [73] |
Kowalski CH, Morelli KA, Schultz D, et al. Fungal biofilm architecture produces hypoxic microenvironments that drive antifungal resistance[J]. Proc Natl Acad Sci U S A, 2020, 117(36): 22473-22483.
[DOI]
|
| [74] |
Lister JL, Horswill AR. Staphylococcus aureus biofilms: recent developments in biofilm dispersal[J]. Front Cell Infect Microbiol, 2014, 4: 178.
|
| [75] |
Wigger C, Leach AJ, Beissbarth J, et al. Povidone-iodine ear wash and oral cotrimoxazole for chronic suppurative otitis media in Australian aboriginal children: study protocol for factorial design randomised controlled trial[J]. BMC Pharmacol Toxicol, 2019, 20(1): 46.
[DOI]
|
| [76] |
Khlebodarova TM, Likhoshvai VA. Persister cells-a plausible outcome of neutral coevolutionary drift[J]. Sci Rep, 2018, 8(1): 14309.
[DOI]
|
| [77] |
Santa Maria PL, Kaufman AC, Bacacao B, et al. Topical therapy failure in chronic suppurative otitis media is due to persister cells in biofilms[J]. Otol Neurotol, 2021, 42(9): e1263-e1272.
[DOI]
|
| [78] |
Costerton JW, Stewart PS, Greenberg EP. Bacterial biofilms: a common cause of persistent infections[J]. Science, 1999, 284(5418): 1318-1322.
[DOI]
|
| [79] |
Alhede M, Bjarnsholt T, Givskov M, et al. Pseudomonas aeruginosa biofilms: mechanisms of immune evasion[J]. Adv Appl Microbiol, 2014, 86: 1-40.
|
| [80] |
Jolivet-Gougeon A, Bonnaure-Mallet M. Biofilms as a mechanism of bacterial resistance[J]. Drug Discov Today Technol, 2014, 11: 49-56.
[DOI]
|
| [81] |
Romling U, Kjelleberg S, Normark S, et al. Microbial biofilm formation: a need to act[J]. J Intern Med, 2014, 276(2): 98-110.
[DOI]
|
| [82] |
Wang JC, Hamood AN, Saadeh C, et al. Strategies to prevent biofilm-based tympanostomy tube infections[J]. Int J Pediatr Otorhinolaryngol, 2014, 78(9): 1433-1438.
[DOI]
|
| [83] |
Khosravi Y, Ling LC, Loke MF, et al. Determination of the biofilm formation capacity of bacterial pathogens associated with otorhinolaryngologic diseases in the Malaysian population[J]. Eur Arch Otorhinolaryngol, 2014, 271(5): 1227-1233.
[DOI]
|
| [84] |
Jurgens G, Lindstrom K, Saano A. Novel group within the kingdom Crenarchaeota from boreal forest soil[J]. Appl Environ Microbiol, 1997, 63(2): 803-805.
[DOI]
|
| [85] |
Watanabe N, Kryukov K, Nakagawa S, et al. Detection of pathogenic bacteria in the blood from sepsis patients using 16S rRNA gene amplicon sequencing analysis[J]. PLoS One, 2018, 13(8): e0202049.
[DOI]
|
| [86] |
Pichler M, Coskun OK, Ortega-Arbulu AS, et al. A 16S rRNA gene sequencing and analysis protocol for the Illumina MiniSeq platform[J]. Microbiologyopen, 2018, 7(6): e00611.
[DOI]
|
| [87] |
Kozich JJ, Westcott SL, Baxter NT, et al. Development of a dual-index sequencing strategy and curation pipeline for analyzing amplicon sequence data on the MiSeq Illumina sequencing platform[J]. Appl Environ Microbiol, 2013, 79(17): 5112-5120.
[DOI]
|
| [88] |
Gosiewski T, Ludwig-Galezowska AH, Huminska K, et al. Comprehensive detection and identification of bacterial DNA in the blood of patients with sepsis and healthy volunteers using next-generation sequencing method - the observation of DNAemia[J]. Eur J Clin Microbiol Infect Dis, 2017, 36(2): 329-336.
[DOI]
|
| [89] |
Gyarmati P, Kjellander C, Aust C, et al. Bacterial landscape of bloodstream infections in neutropenic patients via high throughput sequencing[J]. PLoS One, 2015, 10(8): e0135756.
[DOI]
|
| [90] |
Johnson JS, Spakowicz DJ, Hong BY, et al. Evaluation of 16S rRNA gene sequencing for species and strain-level microbiome analysis[J]. Nat Commun, 2019, 10(1): 5029.
[DOI]
|
| [91] |
Zhao L, Zhang F, Ding X, et al. Gut bacteria selectively promoted by dietary fibers alleviate type 2 diabetes[J]. Science, 2018, 359(6380): 1151-1156.
[DOI]
|
| [92] |
Van Nood E, Vrieze A, Nieuwdorp M, et al. Duodenal infusion of donor feces for recurrent Clostridium difficile[J]. N Engl J Med, 2013, 368(5): 407-415.
[DOI]
|
| [93] |
Guan H, Shen A, Lv X, et al. Detection of virus in CSF from the cases with meningoencephalitis by next-generation sequencing[J]. J Neurovirol, 2016, 22(2): 240-245.
[DOI]
|
| [94] |
Yu G, Zhao W, Shen Y, et al. Metagenomic next generation sequencing for the diagnosis of tuberculosis meningitis: A systematic review and meta-analysis[J]. PLoS One, 2020, 15(12): e0243161.
[DOI]
|
| [95] |
Grumaz S, Stevens P, Grumaz C, et al. Next-generation sequencing diagnostics of bacteremia in septic patients[J]. Genome Med, 2016, 8(1): 73.
[DOI]
|
| [96] |
Crawford E, Kamm J, Miller S, et al. Investigating transfusion-related sepsis using culture-independent metagenomic sequencing[J]. Clin Infect Dis, 2020, 71(5): 1179-1185.
[DOI]
|
| [97] |
Mouraviev V, Mcdonald M. An implementation of next generation sequencing for prevention and diagnosis of urinary tract infection in urology[J]. Can J Urol, 2018, 25(3): 9349-9356.
|
| [98] |
Miao Q, Ma Y, Wang Q, et al. Microbiological diagnostic performance of metagenomic next-generation sequencing when applied to clinical practice[J]. Clin Infect Dis, 2018, 67(suppl_2): S231-S240.
[DOI]
|
| [99] |
Huang ZD, Zhang ZJ, Yang B, et al. Pathogenic detection by metagenomic next-generation sequencing in osteoarticular infections[J]. Front Cell Infect Microbiol, 2020, 10: 471.
[DOI]
|
 2024, Vol. 30
2024, Vol. 30


